ML | Spektrale Clusterbildung
Voraussetzungen: K-Means Clustering
Spectral Clustering ist ein wachsender Clustering-Algorithmus, der in vielen Fällen eine bessere Leistung als viele herkömmliche Clustering-Algorithmen erbracht hat. Es behandelt jeden Datenpunkt als Graphknoten und wandelt so das Clustering-Problem in ein Graph-Partitionierungsproblem um. Eine typische Implementierung besteht aus drei grundlegenden Schritten:
- Erstellen des Ähnlichkeitsdiagramms: In diesem Schritt wird das Ähnlichkeitsdiagramm in Form einer Adjazenzmatrix erstellt, die durch A dargestellt wird. Die Adjazenzmatrix kann auf folgende Arten erstellt werden:
- Epsilon-Nachbarschaftsgraph: Ein Parameter epsilon wird vorher festgelegt. Dann wird jeder Punkt mit allen Punkten verbunden, die in seinem Epsilon-Radius liegen. Wenn alle Abstände zwischen zwei beliebigen Punkten im Maßstab ähnlich sind, werden normalerweise die Gewichte der Kanten, dh der Abstand zwischen den beiden Punkten, nicht gespeichert, da sie keine zusätzlichen Informationen liefern. In diesem Fall handelt es sich bei dem erstellten Diagramm also um ein ungerichtetes und ungewichtetes Diagramm.
- K-Nächste Nachbarn Ein Parameter k ist vorher festgelegt. Dann wird für zwei Eckpunkte u und v eine Kante nur dann von u nach v gerichtet, wenn v zu den k-nächsten Nachbarn von u gehört. Es ist zu beachten, dass dies zur Bildung eines gewichteten und gerichteten Graphen führt, da es nicht immer der Fall ist, dass für jedes u, das v als einen der k-nächsten Nachbarn hat, es der gleiche Fall ist, wenn v u unter seinem k-nächsten hat Nachbarn. Um dieses Diagramm ungerichtet zu machen, wird einer der folgenden Ansätze verfolgt: -
- Direkt eine Kante von u nach v und v u zu , wenn entweder V unter den k-nächsten Nachbarn von u OR u ist unter den k-nächsten Nachbarn von v.
- Richten Sie eine Kante von u nach v und von v nach u, wenn v zu den k-nächsten Nachbarn von u gehört UND u zu den k-nächsten Nachbarn von v gehört.
- Vollständig verbundenes Diagramm: Um dieses Diagramm zu erstellen, wird jeder Punkt mit einer ungerichteten Kante verbunden, die durch den Abstand zwischen den beiden Punkten zu jedem anderen Punkt gewichtet wird. Da dieser Ansatz verwendet wird, um die lokalen Nachbarschaftsbeziehungen zu modellieren, wird typischerweise die Gaußsche Ähnlichkeitsmetrik verwendet, um die Entfernung zu berechnen.
- Projizieren der Daten auf einen niedrigeren Dimensionsraum: Dieser Schritt wird ausgeführt, um die Möglichkeit zu berücksichtigen, dass Mitglieder desselben Clusters im angegebenen Dimensionsraum weit entfernt sind. Somit wird der Dimensionsraum reduziert, so dass diese Punkte näher im reduzierten Dimensionsraum liegen und somit durch einen herkömmlichen Clustering-Algorithmus zusammen geclustert werden können. Dies erfolgt durch Berechnung der Graph Laplace-Matrix . Um dies jedoch zuerst zu berechnen, muss der Grad eines Knotens definiert werden. Der Grad des i-ten Knotens ist gegeben durch
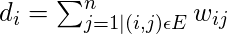
Beachten Sie, dass dies
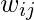 die Kante zwischen den Knoten i und j ist, wie in der obigen Adjazenzmatrix definiert.
die Kante zwischen den Knoten i und j ist, wie in der obigen Adjazenzmatrix definiert.Die Gradmatrix ist wie folgt definiert: -
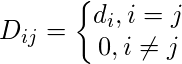
Somit ist die Graph Laplace Matrix definiert als:
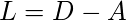
Diese Matrix wird dann für die mathematische Effizienz normalisiert. Um die Dimensionen zu reduzieren, werden zunächst die Eigenwerte und die jeweiligen Eigenvektoren berechnet. Wenn die Anzahl der Cluster k ist, werden die ersten Eigenwerte und ihre Eigenvektoren genommen und in eine Matrix gestapelt, so dass die Eigenvektoren die Spalten sind.
- Clustering der Daten: Dieser Prozess umfasst hauptsächlich das Clustering der reduzierten Daten mithilfe einer herkömmlichen Clustering-Technik - normalerweise K-Means Clustering. Zunächst wird jedem Knoten eine Zeile der normalisierten Graph Laplace-Matrix zugewiesen. Dann werden diese Daten unter Verwendung einer herkömmlichen Technik geclustert. Um das Clustering-Ergebnis zu transformieren, wird die Knoten-ID beibehalten.
Eigenschaften:
- Annahme weniger: Bei dieser Clustering-Technik wird im Gegensatz zu anderen herkömmlichen Techniken nicht davon ausgegangen, dass die Daten einer bestimmten Eigenschaft folgen. Dies macht diese Technik somit zur Beantwortung einer allgemeineren Klasse von Clusterproblemen.
- Einfache Implementierung und Geschwindigkeit: Dieser Algorithmus ist einfacher zu implementieren als andere Clustering-Algorithmen und auch sehr schnell, da er hauptsächlich aus mathematischen Berechnungen besteht.
- Nicht skalierbar: Da Matrizen erstellt und Eigenwerte und Eigenvektoren berechnet werden, ist dies für dichte Datensätze zeitaufwändig.
Die folgenden Schritte zeigen, wie Sie Spectral Clustering mit Sklearn implementieren. Die Daten für die folgenden Schritte sind die Kreditkartendaten, die von Kaggle heruntergeladen werden können .
Schritt 1: Importieren der erforderlichen Bibliotheken
importpandas as pdimportmatplotlib.pyplot as pltfromsklearn.clusterimportSpectralClusteringfromsklearn.preprocessingimportStandardScaler, normalizefromsklearn.decompositionimportPCAfromsklearn.metricsimportsilhouette_score
Schritt 2: Laden und Bereinigen der Daten
cd"C:\Users\Dev\Desktop\Kaggle\Credit_Card"X=pd.read_csv('CC_GENERAL.csv')X=X.drop('CUST_ID', axis=1)X.fillna(method='ffill', inplace=True)X.head()

Schritt 3: Vorverarbeitung der Daten, um die Daten sichtbar zu machen
scaler=StandardScaler()X_scaled=scaler.fit_transform(X)X_normalized=normalize(X_scaled)X_normalized=pd.DataFrame(X_normalized)pca=PCA(n_components=2)X_principal=pca.fit_transform(X_normalized)X_principal=pd.DataFrame(X_principal)X_principal.columns=['P1','P2']X_principal.head()

Schritt 4: Erstellen der Clustering-Modelle und Visualisieren des Clustering
In den folgenden Schritten werden zwei verschiedene Spectral Clustering-Modelle mit unterschiedlichen Werten für den Parameter 'Affinität' erstellt. Informationen zur Dokumentation der Spectral Clustering-Klasse finden Sie hier .
a) Affinität = 'rbf'
spectral_model_rbf=SpectralClustering(n_clusters=2, affinity='rbf')labels_rbf=spectral_model_rbf.fit_predict(X_principal)
colours={}colours[0]='b'colours[1]='y'cvec=[colours[label]forlabelinlabels_rbf]b=plt.scatter(X_principal['P1'], X_principal['P2'], color='b');y=plt.scatter(X_principal['P1'], X_principal['P2'], color='y');plt.figure(figsize=(9,9))plt.scatter(X_principal['P1'], X_principal['P2'], c=cvec)plt.legend((b, y), ('Label 0','Label 1'))plt.show()

b) Affinität = 'nächste Nachbarn'
spectral_model_nn=SpectralClustering(n_clusters=2, affinity='nearest_neighbors')labels_nn=spectral_model_nn.fit_predict(X_principal)

Schritt 5: Bewertung der Leistungen
affinity=['rbf','nearest-neighbours']s_scores=[]s_scores.append(silhouette_score(X, labels_rbf))s_scores.append(silhouette_score(X, labels_nn))(s_scores)
![]()
Schritt 6: Vergleichen der Leistungen
plt.bar(affinity, s_scores)plt.xlabel('Affinity')plt.ylabel('Silhouette Score')plt.title('Comparison of different Clustering Models')plt.show()

